Prosedur RT-qPCR untuk Analisis Ekspresi Gen pada Bakteri
Bagaimana cara melakukan prosedur laboratorium untuk analisis ekspresi gen, khususnya untuk bakteri dalam penelitian bioteknologi dan biomedis.
A. Prinsip Kerja RT-qPCR
Pengujian reverse-transkripsi PCR real-time kuantitatif atau yang disebut RT-qPCR merupakan metode yang menggunakan dye DNA-binding fluoresen dimana kini menjadi metode gold-standard untuk mempelajari ekspresi gen bakteri melalui perhitungan atau kuantifikasi relatif mRNA target tertentu. Metode ini memungkinkan analisis ekspresi gen dalam setiap sel tunggal. Layaknya metode PCR pada umumnya, prosedur RT-qPCR perlu dilakukan secara hati-hati untuk mendapatkan hasil yang reliable, khususnya berkaitan kualitas sampel RNA dan pilihan gen referensi target. Pada kesempatan ini kami akan menjabarkan langkah-langkah kuantifikasi kadar transkrip gen bakteri menggunakan metode RT-qPCR.
Untuk mendapatkan analisis ekspresi gen akurat dengan metode RT-qPCR, terdapat beberapa poin yang harus diperhatikan, diantaranya:
- desain penelitian yang sesuai dan tepat
- kontrol kualitas ekstraksi dan penyimpanan RNA
- kontrol kontaminasi gDNA
- penggunaan gen referensi tervalidasi
- pilihan alur kerja analisis data yang tepat
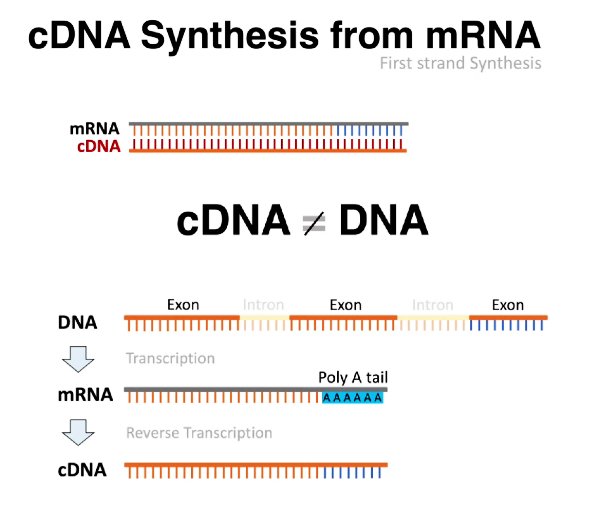
Gambar 1. Sintesis cDNA dari RNA untuk RT-qPCR
B. Metode RT-qPCR
1. Bahan
Reagen yang digunakan harus memiliki grade Molecular Biology, plastic tube (200 μl dan 1,5 ml) harus nuclease-free dan microtip ber-filter (untuk ukuran 10 μl, 100 μl, 1000 μl). Semua bahan dengan karakteristik tersebut difungsikan pada semua langkah ekstraksi, purifikasi, analisis RNA, dan RT-qPCR. Pencegahan kontaminasi RNase harus dilakukan guna mencegah segala kemungkinan hasil non-valid. Pencegahan tersebut dapat dilakukan dengan menggunakan Surface RNase Erasol (Solarbio: SR0040) untuk mengusap semua permukaan dan alat yang digunakan.
Bapak untuk Ekstraksi dan Purifikasi RNA:
- TE Buffer (pH 8.0) (Solarbio: T1120), opsional: 15 mg/ml lysozyme (untuk lisis bakteri).
- RNAwait (Solarbio: SR0020)
- EasySC Universal RNA Purification Kit (ABPbio: R137-1)
- Kloroform grade Molecular Biology
- Etanol absolut
- Water, DEPC treated, RNase tested (Nacalai: 36420-61)
- Opsional: SV Total RNA Isolation System kit (Promega)
- Opsional: RNase H atau Ribonuclease H (Solarbio:R7900)
- Microcentrifuge Refrigerated
- Instrumentasi qPCR Machine: Real-Time PCR Detection System
Bahan untuk Reverse Transkripsi and qPCR:
- iScript™ IV First-Strand cDNA Synthesis Kit (ABPBio: D041)
- 2×Taq PCR Master Mix with Dye (ABPBio: D009)
- Barcoded 96-well PCR plate (atau 8-strip PCR tube)
- Microseal ‘B’ PCR Plate Sealing Film
- Instrumentasi qPCR Machine: Real-Time PCR Detection System
2. Isolasi dan Pemurnian RNA
1) Isolasi RNA
- Tempatkan 500 μl kultur bakteri ke dalam tube 2 ml baru dan tambahkan 1 ml RNAwait (Solarbio: SR0020) atau RNAprotect Bacteria Reagent (Qiagen). Vorteks kultur secara sempurna dan inkubasi selama 5 menit pada suhu ruang.
- Sentrifugasi tube pada 10.000 ×g selama 10 menit. Buang supernatan dengan melakukan inversi dan lanjutkan ke langkah selanjutnya. Apabila ingin melakukan penyimpanan hingga 1 bulan, pelet dapat disimpan pada suhu -70°C.
Beberapa langkah yang perlu diperhatikan sebelum melakukan purifikasi
- Buffer RL dapat membentuk endapan selama penyimpanan. Direkomendasikan untuk menghangatkan hingga 50°C agar larut dengan sempurna.
- Buffer AW2 disediakan dalam bentuk konsentrat, sebelum digunakan perlu ditambahkan etanol absolut dalam jumlah yang sesuai (96-100%) seperti dideskripsikan pada label botol untuk memperoleh larutan kerja.
- Larutan kerja Lisozim dibuat dengan melarutkan lisozim (Sigma-Aldrich) 10 mg/mL dalam TE Buffer (pH 8.0) (Solarbio: T1120). Lisozim bertujuan untuk melisiskan dinding sel bakteri.
- Homogenisasi sampel bakteri dimulai dengan melakukan sentrifugasi untuk mengoleksi bakteri (<1×108 sel), tambahkan 100 μl larutan kerja Lisozim dan inkubasi pada suhu ruang selama 10 menit. Kemudian tambahkan 1 mL Buffer RL dan vorteks secara menyeluruh selama 1 menit.
- Inkubasi campuran pada suhu ruang selama 5-10 menit.
- Tambahkan 400 μl ddH2O dan kocok dengan kuat menggunakan tangan selama 15 detik dan inkubasi pada suhu ruang selama 5-15 menit. Kocokan harus dilakukan secara cepat dan kuat. Pencampuran lambat akan menghasilkan ekstraksi tidak optimal. Vorteks harus dihindari karena akan menyebabkan kontaminasi DNA. Penambahan ddH2O harus sesuai proporsi, apabila terlalu banyak ddH2O akan mengembalikan DNA dan protein ke fase air sehingga menurunkan kemurnian RNA.
- Sentrifugasi campuran dilakukan pada 12.000 ×g selama 15 menit pada suhu ruang. Setelah sentrifugasi dilakukan, Komponen DNA, protein dan polisakarida membentuk endapan di bagian bawah, sedangkan RNA tetap berada di supernatan.
- Transfer supernatan ke dalam tube 2 mL baru, kemudian tambahkan 0,5 volume etanol absolut dan vorteks selama 10 detik. Untuk memperoleh small RNA, tambahkan 1,5 volume etanol absolut ke supernatan.
- Hubungkan RNA Mini Column ke tube koleksi 2 mL. Transfer 600 μl sampel dari langkah 7 ke dalam RNA Mini Column dan sentrifugasi pada 12.000 ×g selama 1 menit.
- Buang filtrat dan gunakan kembali tube koleksi.Transfer sisa sampel dari langkah 7 ke RNA Mini Column dan sentrifugasi pada 12.000 ×g selama 1 menit.
- Buang filtrat dan gunakan kembali tube koleksi. Tambahkan 600 μl Buffer AW2 ke RNA Mini Column, kemudian disentrifugasi pada 12.000 ×g selama 1 menit. Buffer AW2 harus diencerkan dengan etanol absolut sesuai keterangan label botol sebelum digunakan.
- Buang filtrat dan gunakan kembali tube koleksi dan sentrifugasi RNA Mini Column yang kosong pada 12.000 ×g selama 3 menit. Langkah ini bersifat penting untuk menghilangkan sisa etanol yang dapat mengganggu aplikasi downstream.
- Transfer RNA Mini Column ke dalam tube 1,5 ml bersih. Tambahkan 30-100 μl DEPC-treated water ke bagian tengah membran silika dari column. Tutup lid dan inkubasi pada suhu ruang selama 3 menit, kemudian disentrifugasi pada 12.000 ×g selama 1 menit. Untuk memperoleh hasil lebih optimal, ulangi langkah ini untuk langkah elusi kedua.
- Buang column dan simpan RNA pada suhu -80ºC.
2) RNA Clean-up (Opsional)
Prosedur pembersihan RNA (RNA Clean-up) direkomendasikan untuk menghilangkan sisa DNA genom (gDNA) atau kontaminan lain dalam total sampel RNA. Prosedur dapat dilakukan menggunakan SV Total RNA Isolation System Kit (Promega).
3. Susunan RT-qPCR
1) Reverse-Transkripsi
Pengujian RT-PCR harus dilakukan dalam lingkungan nuclease-free untuk memaksimalkan hasil yang diperoleh. Penggunaan mikropipet nuclease-free dan microtips dengan filter sangat direkomendasikan untuk mencegah segala kemungkinan kontaminasi.
- Proses thawing template RNA dan reagen harus dilakukan atas blok es dingin. Vorteks semua larutan reagen untuk homogenisasi dan sentrifugasi dalam waktu singkat untuk mengoleksi sisa cairan dari bagian sisi tube.
- Siapkan campuran reaksi berikut dalam tube PCR di atas blok es:
- RNA total (atau poly(A)+RNA) (dalam µl),
- 2 µl 2× RT Reaction Mix
- 18 µl Nuclease-free H2O.
- Konsentrasi akhir RNA: 1 ng – 2 µg total RNA atau 10 pg – 500 ng mRNA. Untuk campuran berikut ditujukkan untuk reaksi ukuran 20 µl (1×).
- Opsional: Hangatkan campuran reaksi hingga 65°C selama 5 menit dan inkubasi di atas blok es setidaknya selama 1 menit. Koleksi semua komponen dengan melakukan sentrifugasi dalam waktu singkat.
- Tambahkan 2 µl RT Enzyme Mix (10×), vorteks selama 3 – 5 detik secara merata dan hati-hati. Sentrifugasi dalam waktu singkat untuk mengoleksi seluruh isi tube.
- Inkubasi tube pada 25°C selama 10 menit, kemudian dilanjutkan pada 50°C selama 50 menit.
- Hentikan reaksi dengan memanaskan pada 85°C selama 5 menit. Dinginkan di atas blok es. First-strand cDNA sintetik baru dapat digunakan langsung untuk PCR.
Catatan:
- Isolasi poli(A)+RNA dari total RNA bersifat opsional dan tidak diharuskan. Namun, proses tersebut dapat meningkatkan hasil dan kemurnian produk akhir. Gunakan NEBNext® Poly(A) mRNA Magnetic Isolation Module (New England Biolabs: E7490).
- Hasil cDNA sintetik umumnya dapat langsung digunakan sebagai template PCR tanpa perlu pemurnian lebih lanjut. Direkomendasikan untuk melakukan pengenceran campuran reaksi akhir sebanyak 10 kali dengan nuclease-free water, baru kemudian gunakan 1 – 2 μl campuran encer tersebut untuk PCR.
- Untuk menghilangkan RNA komplementer terhadap cDNA, tambahkan 1 μl (2 Unit) E. coli RNase H (Ribonuclease H, Solarbio:R7900) dan inkubasi pada 37°C selama 20 menit.
- Sampel RNA yang digunakan harus bebas dari kontaminasi DNA genom (gDNA). Gunakan SV Total RNA Isolation System kit (Promega) untuk menghilangkan kontaminasi gDNA.
- Berbeda dengan priming oligo(dT) yang biasanya tidak memerlukan optimasi, rasio random primer terhadap RNA sangat penting hubungannya dengan panjang rata-rata cDNA sintetik dalam reaksi. Peningkatan rasio random primer/RNA akan menghasilkan hasil cDNA pendek (~500bp) yang lebih banyak, sedangkan penurunan rasio tersebut akan menghasilkan produk lebih panjang.
- Produk cDNA sintetik harus disimpan pada suhu -20°C.
2) Setup qPCR
- Rancang dan print template well-plate sesuai dengan well-plate qPCR pilihan. Gunakan template tersebut untuk mengidentifikasi sumuran dan merencanakan distribusi sampel di seluruh well-plate qPCR. Setiap templat cDNA dan reaksi kontrol negatif harus dipreparasi setidaknya dalam duplikat.
- Proses thawing qPCR Master Mix dilakukan pada suhu ruang dan vorteks secara perlahan, kemudian sentrifugasi dalam waktu singkat untuk menghindari busa dan paparan cahaya. Simpan reagen di dalam es hingga akan digunakan.
- Dalam tube steril baru, preparasi qPCR reaction mix menggunakan reagen dengan urutan sebagai berikut:
- 2×Taq PCR Master Mix (ABPBio);
- 2 μM forward primer;
- 2 μM reverse primer;
- nuclease-free water (sampai volume akhir 16 μl)
- Transfer 16 μl qPCR Reaction Master Mix ke sumuran well-plate dengan hati-hati sesuai dengan rancangan print-out pada langkah 1. Jagalah well-plate tetap pada suhu ruang.
- Transfer 4 μl templat cDNA ke sumuran secara hati-hati. Gunakan 4 μl nuclease-free water untuk preparasi reaksi ‘no-template control’ (NTC)..
- Tutup well-plate secara hati-hati menggunakan optical adhesive. Berikan tekanan pada ruang di antara sumur menggunakan benda plastik bersih dan tajam. Pastikan sumur tertutup rapat.
- Sentrifugasi well-plate dengan kecepatan rendah selama 1 menit untuk mengkumpulkan volume reaksi dan menghilangkan gelembung.
- Tempatkan well-plate qPCR dengan hati-hati pada blok Platform PCR Machine
- Atur program siklus sebagai berikut:
- 1 siklus aktivasi polimerase pada suhu 95°C selama 2 menit;
- 40 siklus denaturasi pada suhu 95°C selama 15 detik,
- annealing/ekstension pada suhu 60°C selama 60 detik;
- kurva disosiasi titik akhir pada 60–95°C.
- Pastikan file data disimpan dengan benar dan mulai running. Opsi deteksi fluoresensi dan analisis melting curve harus diaktifkan pada thermal cycler tersebut. Pengelolaan reaksi qPCR dan analisis hasil qPCR dapat dilakukan dengan software REST (Qiagen), CFX Maestro (Bio-rad) atau lain sebagainya.
C. Analisis Efisiensi Primer dan Ekspresi Gen
1. Determinasi Efisiensi Primer
Efisiensi amplifikasi set primer dalam reaksi qPCR bergantung langsung pada sekuens oligonukleotida. Efisiensi harus ditentukan secara eksperimental untuk setiap set primer dengan menyiapkan pengenceran serial sepuluh kali lipat dari lima titik untuk sampel cDNA tertentu (yaitu, 100, 10-1, 10-2, 10-3, 10-4). Efisiensi amplifikasi dapat ditentukan menggunakan alat online khusus, qPCR Efficiency Calculator (website Thermo Fisher).
2. Determinasi Efisiensi Primer
Beberapa author publikasi ilmiah merekomendasikan untuk melakukan seleksi gen referensi kandidat untuk bakteri berdasarkan meta-analisis data ekspresi gen dari platform Pathosystems Resource Integration Center (www.patricbrc.org).
3. Penilaian Stabilitas Ekspresi Gen
Penilaian stabilitas ekspresi gen dapat dilakukan dengan menggunakan program add-in NormFinder yang dapat diunduh untuk program Microsoft Excel® a (www.moma.dk/normfinder-software)
D. Referensi
- Domingues L. 2023. PCR: Methods and Protocols. 2nd Ed. Humana. Springer.
- Domingues L. 2017. PCR: Methods and Protocols. 1st Ed. Humana. Springer.
- Bartlett JMS, Stirling D. 2003. PCR Protocols. Humana. Springer.
- Biassoni R, Raso A. 2020. Quantitative Real-Time PCR: Methods and Protocols. Humana. Springer.
- Wilks M. 2013. PCR Detection of Microbial Pathogens. Humana. Springer.
Artikel terkait
- Deteksi Infeksi Patogen dalam Transplantasi Organ dan Jaringan dengan Liferiver Real-Time PCR Kit
- Skrining Kanker Menggunakan Kit Real-Time PCR Onkologi Generi Biotech
- Diagnosis Mutasi Trombofilia Menggunakan Kit Real-time Pcr Dari Generi-biotech
- Deteksi Penyakit Pada Tanaman Dengan PCR Kit Merk Loewe Germany
- PCR Kit IVD untuk deteksi SARS-CoV-2
- Deteksi Adulterant dan Kontaminan Pada Pangan Dengan Metode ELISA, PCR dan Rapid Test
- Real Time PCR, Level Baru Amplifikasi DNA
- Tahukah Anda tentang Conventional PCR / PCR Konvensional
- Mengenal Plasmid dalam Penelitian Genetik

