Workshop bag. 1 : Pelatihan Proteomic Assay di Fakultas Kedokteran Gigi Universitas Indonesia
Departemen Biologi Oral Fakultas Kedokteran Gigi Universitas Indonesia mengadakan pelatihan dan workshop yang memiliki fokus materi Biomolecular Techniques Proteomic Assay. Workshop dilaksanakan pada tanggal 17-18 Juli 2024 di Laboratorium Oral Biologi – Fakultas Kedokteran Gigi Universitas Indonesia, Jl. Salemba Raya, Kenari, Senen, Kota Jakarta Pusat, DKI Jakarta 10430. Dalam pelaksanaan workshop tersebut FKG UI berkolaborasi dengan PT. Indogen Intertama.
Dalam penelitian proteomik akan melibatkan berbagai teknik analisis seperti ELISA, SDS-PAGE, Western Blot, dan In Silico, biasanya urutan analisis tersebut bergantung pada tujuan spesifik penelitian dan data yang diperlukan pada setiap tahap.
Bag 1 – Analysis In Silico
Studi in silico adalah uji yang dilakukan menggunakan komputer dan perangkat lunak (analisis bioinformatika), studi ini dilakukan sebagai pendekatan alternatif yang terus dikembangkan untuk membatasi, mengurangi, melakukan prediksi aktifitas dan sifat molekul obat sebelum dilakukan pengujian lebih lanjut di laboratorium. Analisis bioinformatika dengan program supercomputer ini menawarkan metode in silico sebagai komplemen metode in vitro dan in vivo yang lazim digunakan dalam proses penemuan obat.
Penyampaian materi Analysis In Silico disampaikan oleh Prof. drg. Endang Winiati Bachtiar, M.Biomed., Ph.D., PBO dan Bapak Turmidzi Fath, M.Sc., M.Si dari Fakultas Kedokteran Gigi Universitas Indonesia.
Analisis in silico dalam penelitian genomik dan proteomik sering kali melibatkan simulasi penambatan molekul atau yang dikenal dengan molecular docking. Molecular docking bertujuan untuk memprediksi cara interaksi dan kekuatan afinitas antara dua molekul. Hasil dari kekuatan interaksi afinitas antara dua molekul menghasilkan skor yang menggambarkan energi total ikatan protein-ligand. Jadi, molecular docking adalah metode berbasis struktur in silico yang banyak digunakan dalam membantu memprediksi interaksi yang terjadi antara molekul dan target biologis. Metode komputasi berbasis struktur ini mampu mengidentifikasi suatu senyawa memiliki potensi sebagai kandidat obat baru berdasarkan informasi dari struktur 3D target (protein) yang berikatan dengan suatu molekul kecil (ligand).
a. Apa saja yang diperlukan untuk molecular docking?
Beberapa preparasi yang penting diperlukan untuk molecular docking diantaranya, protein, ligand, dan asam nukleat.
- Reseptor adalah protein dengan binding pocket.
Komponen yang paling penting dalam reseptor obat adalah protein. Protein target adalah molekul biologis yang menjadi sasaran interaksi ligan. Protein target yang biasa digunakan yaitu enzim, reseptor, atau protein lainnya yang terlibat dalam jalur biologis tertentu.
- Ligand merupakan senyawa yang akan menempel pada binding pocket.
Ligan adalah molekul kecil yang akan berinteraksi dengan target protein. Ligan dapat berupa obat, senyawa kimia, atau molekul alami.
- Asam nukleat (jika relevan dengan penelitian)
Dalam beberapa penelitian, target docking bisa berupa DNA atau RNA. Ini relevan dalam penelitian yang melibatkan interaksi ligan dengan materi genetik.
b. Tipe molecular docking
Ada beberapa tipe molecular docking yang digunakan untuk berbagai tujuan dalam penelitian, seperti berikut :
- Rigid body docking, dimana reseptor bersifat kaku dan ligan bersifat kaku.
- Flexible ligand docking, dimana reseptor bersifat kaku dan ligan bersifat fleksibel atau digerakkan ke segala arah.
- Flexible docking, dimana fleksibilitas reseptor dan ligan dapat dipertimbangkan atau dibuat fleksibel keduanya.
- Blind docking, dilakukan tanpa mengetahui lokasi pasti dari situs aktif pada protein. Ligan dicari di seluruh permukaan protein untuk menemukan situs pengikatan potensial.
- Site-specific docking, fokus pada situs aktif yang sudah diketahui atau diprediksi dalam protein berdasarkan penelitian sebelumnya.
- Oriented docking, sebuah pendekatan dalam molecular docking dimana ligan ditempatkan dalam situs aktif protein dengan orientasi awal tertentu, berdasarkan penelitian sebelumnya. Setelah orientasi awal ditentukan, proses docking dilakukan untuk mengoptimalkan interaksi antara ligan dan protein.
c. Uji Molecular Dynamic Simulation
Molecular Dynamic Simulation (MDS) adalah teknik yang digunakan untuk mempelajari perilaku dinamis dari sistem biomolekuler, seperti protein, ligand, dan kompleks protein-ligand, dalam jangka waktu tertentu.
Molecular docking biasanya memperlakukan protein dan ligan sebagai entitas kaku atau dengan fleksibilitas terbatas. Pengujian MDS membantu mengetahui pergerakan dan interaksi yang terjadi secara real time selama waktu simulasi (misalnya dalam waktu 0-200 nanosecond).
d. Tahapan molecular docking
Secara garis besar, tahapan dalam molecular docking adalah (1) pemilihan dan preparasi reseptor & ligand, (2) proses molecular docking, (3) visualisasi molekul dan analisis data.
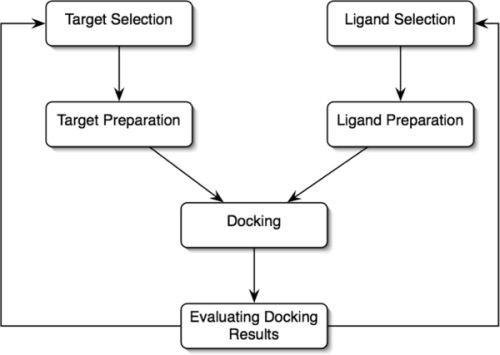
Gambar 1. General workflow analysis Molecular Docking
- Preparasi Reseptor dan Ligan
Struktur 3D dari protein dapat diunduh dari Protein Data Bank dengan format .pdb. Protein Data Bank adalah satu satunya penyedia dan penyimpan informasi berupa struktur 3D protein, asam nukleat dan struktur kompleks RCSB PDB (Research Collaboratory for Structural Bioinformatics Protein Data Bank) dan dapat diakses pada link berikut : https://www.rcsb.org/.pdb . Protein perlu dilakukan pembersihan struktur dengan menghapus molekul air, ion, dan ligan asli yang tidak relevan dari struktur protein. Kemudian menambahkan atom hidrogen yang diperlukan untuk simulasi yang akurat. Dan penentuan situs aktif sebagai tempat pengikatan ligan.Sedangkan struktur 3D dari ligand dapat diunduh dari database seperti PubChem. PubChem merupakan database kimia terbuka pada National Institute of Health (NIH). Molekul yang ada di PubChem sebagian besar adalah molekul kecil dan juga besar yaitu senyawa obat, nukleotida, karbohidrat, lipid, peptida dan makromolekul modifikasi.Ligan juga diperlukan penambahan atom hidrogen (H) untuk memastikan struktur yang lengkap. Penambahan atom H berfungsi pada saat penambatan yang akan membentuk interaksi molekuler yaitu ikatan hidrogen.
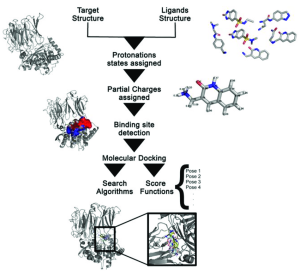
Gambar 2. Preparasi Protein Target dan Ligand
- Proses Molecular Docking
Proses docking ini dilakukan setelah melalui tahap preparasi ligan dan reseptor sehingga ligan dan reseptor siap digunakan. Pada proses running molecular docking ini menggunakan software tertentu. Prinsip dalam penambatan yaitu menambatkan ligan pada bagian aktif reseptor sehingga terjadi interaksi yang dapat dianalisis. - Simulasi Molecular Dynamic Solution
Kelebihan simulasi MSD adalah dapat ditambahkannya molekul pelarut air maupun pelarut organik secara eksplisit pada sistem. Simulasi dinamika molekuler pada sistem pelarut ini mungkin akan diaplikasikan pada ekstraksi di laboratorium, sehingga dengan dilakukannya simulasi ini, diharapkan dapat mengetahui kestabilan, pergerakan, dan distribusi kompleks protein-ligand. Struktur awal menggunakan struktur protein-ligan yang diperoleh dari hasil docking.Durasi simulasi biasanya berkisar antara beberapa nanodetik (ns) hingga mikrodetik (μs) tergantung pada kebutuhan penelitian dan kompleksitas.Parameter yang dianalisis dalam Molecular Dynamic Simulation diantaranya :
-
- Root Mean Square Deviation (RMSD), Nilai RMSD yang konstan setelah beberapa waktu menunjukkan stabilitas struktur. Fluktuasi besar dapat mengindikasikan perubahan konformasi atau ketidakstabilan.
- Root Mean Square Fluctuation (RMSF), residu nilai RMSF tinggi menunjukkan area yang lebih dinamis atau fleksibel dalam protein. Ini bisa menunjukkan loop, heliks, atau daerah yang tidak terstruktur.
- Radius of Gyration (RG), nilai RG yang konstan menunjukkan bahwa protein atau kompleks tetap kompak selama simulasi. Perubahan besar dalam RG dapat menunjukkan unfolding atau perubahan struktural besar.
- Visualisasi Interaksi Molekul
Visualisasi molekul dilakukan untuk mengetahui interaksi antara ligan dan protein. Interaksi yang sering terjadi pada ligan-protein berupa ikatan hidrogen, interaksi hidrofobik, dan gaya van der Waals. Selanjutnya pilih ligand-interaction untuk menampilkan visualisasi dalam bentuk 2D atau 3D.
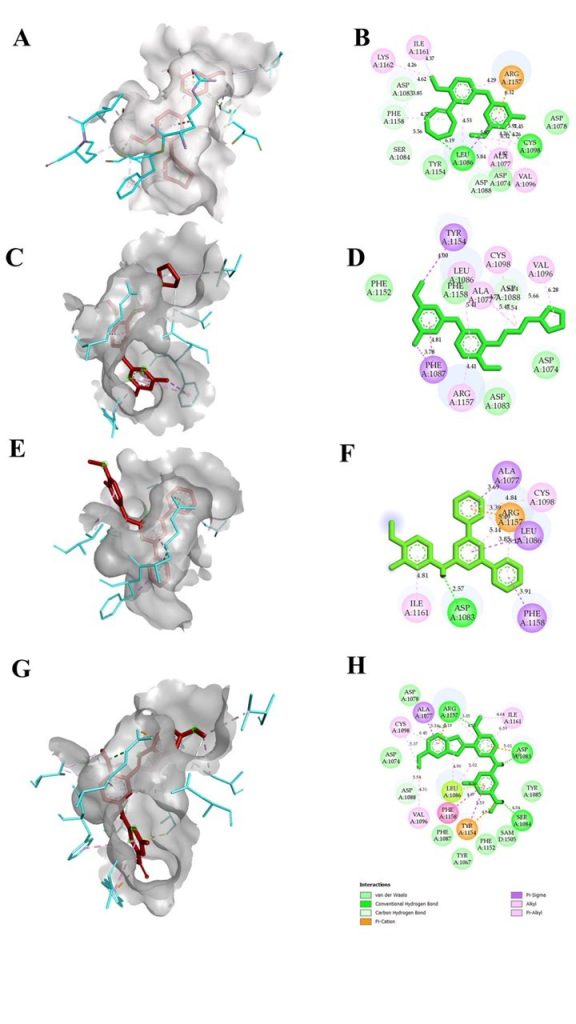
Gambar 3. Contoh visualisasi interaksi molekuler melalui software Autodock dalam tampilan 3D dan 2D pada penelitian : In Silico Molecular Docking and Dynamics Simulation Analysis of Potential Histone Lysine MethylTransferase Inhibitors for Managing β-Thalassemia
- Analisis Data dan Validasi Docking
Hasil dari penambatan molekul pada penelitian ini yang akan dianalisis meliputi energi bebas Gibbs (kcal/mol), nilai konstanta inhibisi, ikatan hidrogen dan kemiripan residu asam amino dari masing-masing ligan uji. Analisis data ini meliputi energi paling stabil serta jenis ikatan dari ligan dan reseptor yang terkuat.Parameter pengujian Molecular Dynamic Solution seperti RMSD, RMSF, dan RG adalah parameter penting untuk mengevaluasi perubahan struktural, fleksibilitas, dan stabilitas keseluruhan selama simulasi. Analisis ini memastikan bahwa hasil molecular docking tidak hanya teoritis tetapi juga valid dalam kondisi dinamis yang lebih realistis.
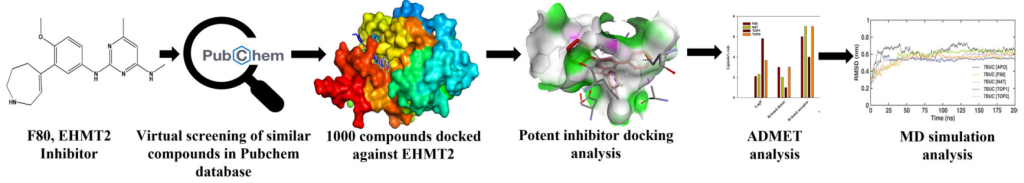
Gambar 4. Ringkasan Analysis In Silico molecular docking dan molecular dynamic simulation pada penelitian : In Silico Molecular Docking and Dynamics Simulation Analysis of Potential Histone Lysine MethylTransferase Inhibitors for Managing β-Thalassemia
e. Software Pendukung Molecular Docking
Beberapa software untuk mendukung penelitian terkait In Silico Analysis yaitu AutoDock Vina untuk docking dan GROMACS atau AMBER untuk uji MSD, bersama dengan alat visualisasi seperti PyMOL, Chimera, dan Visual Molecular Dynamics (VMD). Terdapat beberapa software tambahan untuk mengetahui score function pada hasil docking. Nilai score mencerminkan energi pengikatan atau afinitas pengikatan antara ligan dan protein. Skor ini membantu dalam mengidentifikasi konformasi terbaik dari ligan yang berikatan dengan target protein.
Analisis In Silico merupakan contoh nyata bagaimana perkembangan teknologi informasi telah merevolusi dunia penelitian ilmiah. Teknologi In Silico memungkinkan para peneliti untuk menjelajahi ide-ide baru. Inovasi ini membuatnya menjadi area yang menarik untuk dieksplorasi lebih lanjut.
Referensi :
Ravikumar, Y., Koonyosying, P., Srichairatanakool, S., Ponpandian, L. N., Kumaravelu, J., & Srichairatanakool, S. (2023). In Silico Molecular Docking and Dynamics Simulation Analysis of Potential Histone Lysine MethylTransferase Inhibitors for Managing β-Thalassemia. Molecules, 28(21), 7266. https://doi.org/10.3390/molecules28217266

